R을 활용한 바이오인포매틱스 [바이오 의료와 생명과학 정보 분석 Bioinformatics]
- 원서명Bioinformatics with R Cookbook (ISBN 9781783283132)
- 지은이포루쉬 프라빈 신하(Paurush Praveen Sinha)
- 옮긴이염현식
- ISBN : 9788960776876
- 33,000원
- 2015년 04월 13일 펴냄 (절판)
- 페이퍼백 | 404쪽 | 188*235mm
- 시리즈 : 데이터 과학, acorn+PACKT
판매처
- 현재 이 도서는 구매할 수 없습니다.
책 소개
요약
생명정보학이라고 일컫는 바이오인포매틱스(Bioinformatics)는 생물학적 데이터를 이용해 여러 분석 과정을 시행하고, 이를 통해 새로운 내용을 확인하는 학문이며, 최근 이러한 생명정보학 분석 과정에 가장 많이 사용되는 언어는 R이다. 이 책은 R을 이용한 다양한 예제를 제공해 생명정보학 분석 과정을 체계적이고 직관적으로 설명한다. 이 책을 통해 바이오인포매틱스, 즉 생명정보학의 이론적 배경뿐 아니라 실제적 접근 방식에 대한 충분한 지식과 기술을 얻을 수 있다.
이 책에서 다루는 내용
■ 웹 페이지를 이용하지 않고 R을 통해 생물학적 데이터 추출
■ 데이터의 주석화, 농축화, 식별자의 전환
■ PubMed를 통해 적절한 문서 확인
■ 종들 간의 계통적 연관성 확인
■ 전장유전체 연관 분석을 통해 유전자 정보와 질환의 연관성 확인
■ 생물학적 또는 임상적 특징에 기반을 둔 환자 분류
■ 출판과 발표에 이용할 수 있는 시각화를 통한 생물학적 데이터의 제시
이 책의 대상 독자
사람들은 가끔 큰 불안감을 가지고 프로그래밍에 접근할 때가 있다. 이 책의 목적은 과학자들에게 생명정보학과 컴퓨터 생명공학에서의 다양한 일반적인 문제점들에 대한 안내를 제공해주는 것이다. 또한 R이 아닌 다른 언어에 익숙한 생명정보학과 컴퓨터 생명공학의 프로그래머들에게 도움을 주고자 한다.
독자들에게는 생명정보학의 기본적인 부분들에 대한 친밀감과 컴퓨터 프로그래밍에 대한 기본 지식이 요구된다. 그러나 있을지 모를 어느 정도의 간극을 메우기 위해 모든 장의 시작 부분에 짧은 기본적인 설명을 제시했다.
이 책은 R을 이용한 기본적 프로그래밍이나 생명정보학과 통계학의 기본에 관한 책이 아니다. 필요할 때마다 연관성이 있는 참고 문헌, 책, 블로그의 제시를 통해 적절한 이론적 내용을 제공했다. 예제는 대부분 바로 사용할 수 있지만, 이에 따라서 분석을 하기 전에 독자가 직접 데이터를 확인해보고 개인적인 느낌을 가져볼 것을 강력히 추천한다.
이 책의 구성
1장, ‘R을 이용한 생명정보학 시작’에서는 R의 기본 작업을 설명한다. 주요 주제는 패키지 설치, 데이터 처리와 조작이다. 대부분의(특히 생명의학) 연구에서 첫 번째 단계인 문헌 탐색을 위한 예제로 내용이 확장된다.
2장, ‘Bioconductor 소개’에서는 Bioconductor에서 패키지를 이용해 기본적인 생명정보학의 문제들, 특히 생물학의 메타데이터와 연관된 문제들을 해결하기 위한 예제를 제공한다. 유전자와 단백질의 ID 전환 및 기능적 농축(functional enrichment)에 관련된 내용을 설명한다.
3장, ‘R을 이용한 서열 분석’에서는 문자 단위의 서열 데이터에 대해 주로 설명한다. 서열 데이터 추출, 서열 정렬, 서열의 패턴 탐색 예제를 다룬다.
4장, ‘R을 이용한 단백질 구조 분석’에서는 배열과 구조 수준에서 단백질을 다루는 방법을 설명한다. 서열이나 구조 분석 같은 단백질 생명정보학의 중요한 관점과 방법을 다룬다. 예제는 단백질 구조 분석, 도메인 주석, 단백질의 구조적 특성 분석 등을 포함한다.
5장, ‘R을 이용한 마이크로어레이 데이터 분석’에서는 마이크로어레이 데이터를 읽고 올리는 내용의 예제로 시작해, 이 데이터의 전처리, 필터링, 마이닝, 기능적 농축의 설명으로 이어진다. 마지막으로 유전자 간의 관계를 나타내기 위한 방법으로 공발현(co-expression) 네트워크를 소개한다.
6장, ‘전장유전체 연관 분석(GWAS) 데이터 분석’에서는 생물학적 추론을 위한 전장유전체 연관 분석 데이터의 분석에 관해 이야기한다. 또한 단위 반복 변이(CNV) 데이터뿐만 아니라 다중 연관 분석에 대해 설명한다.
7장, ‘질량 분석 데이터 분석’에서는 질량 분석 데이터의 다양한 관점을 다룬다. 서로 다른 데이터 형식을 읽어 들이고 분석하고 수량화하는 주제를 다룬다.
8장, ‘차세대 시퀀싱 데이터 분석’에서는 차세대 시퀀싱 데이터 처리, RNA 시퀀싱, 칩 시퀀싱, 메틸화 데이터를 다루는 예제에 대해 설명한다.
9장, ‘생명정보학에서의 기계 학습’에서는 생명정보학에서의 기계 학습에 관한 예제를 다룬다. 생물학적 데이터로부터의 추론을 위한 군집화, 분류, 베이지안 학습에 대한 주제를 다룬다.
부록 A, ‘R에서 유용한 연산자와 함수’에서는 다양한 일반적이거나 비일반적인 연산을 시행할 수 있는 유용한 R 함수에 대해 설명한다.
부록 B, ‘유용한 R 패키지’에서는 여러 다른 유형의 분석과 시각화를 위한 유틸리티를 담고 있는 흥미로운 라이브러리들의 목록과 설명을 제공한다.
본문에 쓰인 컬러 이미지는 여기에서 내려 받으세요.
목차
목차
- 1장 R을 이용한 생명정보학 시작
- 소개
- 시작과 라이브러리 설치
- 데이터 읽고 쓰기
- 데이터 필터링과 세분화
- 데이터에 대한 기본적인 통계 작업
- 확률 분포 생성
- 데이터에 대한 통계적 검정 시행
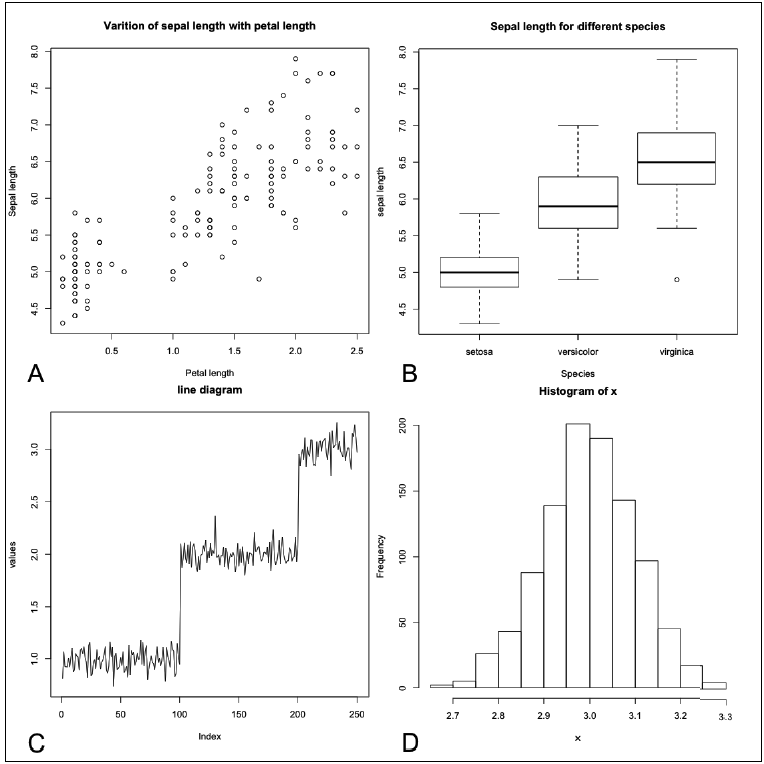
- 데이터 시각화
- R에서 PubMed를 이용해 작업
- BioMart로부터 데이터 검색
- 2장 Bioconductor 소개
- 소개
- Bioconductor로부터 패키지 설치
- R에서 주석 데이터베이스 다루기
- ID 전환
- 유전자 KEGG 주석
- 유전자 온톨로지(GO) 주석
- GO 농축
- 유전자 KEGG 농축
- 클라우드에서의 Bioconductor
- 3장 R을 이용한 서열 분석
- 소개
- 서열 추출
- FASTA 파일 읽기와 쓰기
- 서열 구성의 내용 확인
- 짝 서열 정렬
- 다중 서열 정렬
- 계통 분석과 트리 플로팅
- BLAST 결과 다루기
- 서열에서 패턴 확인
- 4장 R을 이용한 단백질 구조 분석
- 소개
- UniProt에서 서열 추출
- 단백질 서열 분석
- 단백질 서열 특성 계산
- PDB 파일 다루기
- InterPro 도메인 주석으로 작업
- Ramchandran 플롯의 이해
- 유사한 단백질 탐색
- 단백질의 이차 구조 특성을 이용한 작업
- 단백질 구조의 시각화
- 5장 R을 이용한 마이크로어레이 데이터 분석
- 소개
- CEL 파일 읽기
- ExpressionSet 객체 만들기
- AffyBatch 객체 다루기
- 데이터의 질 확인
- 인위적 발현 데이터 생성
- 데이터 정규화
- 발현 데이터에서 배치 효과 해결
- 주성분 분석을 이용한 탐색적 분석
- 차별 발현 유전자 확인
- 다중 클래스 데이터로 작업
- 시계열 데이터 다루기
- 마이크로어레이 데이터의 배율 변화
- 데이터의 기능적 농축
- 마이크로어레이 데이터 군집화
- 마이크로어레이 데이터로부터 공발현 네트워크 형성
- 유전자 발현 데이터의 시각화
- 6장 전장유전체 연관 분석(GWAS) 데이터 분석
- 소개
- 단일 염기 다형성 연관 분석
- 단일 염기 다형성에 대한 연관성 탐색
- 전체 유전체 단일 염기 다형성 연관 분석
- PLINK 전장유전체 연관 분석 데이터 불러오기
- GWASTools 패키지를 이용해 데이터 다루기
- 다른 전장유전체 연관 분석 데이터 형식 다루기
- 단일 염기 다형성 주석화와 농축
- Hardy-Weinberg 평형에 대한 데이터 검정
- 단위 반복 변이 데이터를 이용한 연관 분석
- 전장유전체 연관 분석의 시각화
- 7장 질량 분석 데이터 분석
- 소개
- mzXML/mzML 형식의 MS 데이터 읽기
- Burker 형식의 MS 데이터 읽기
- mzXML 형식의 MS 데이터를 MALDIquant 형식으로 전환
- MS 데이터 객체로부터 데이터 요소 추출
- MS 데이터 전처리
- MS 데이터에서 피크 검출
- MS 데이터의 피크 정렬
- MS 데이터에서 펩타이드 확인
- 단백질 양적 분석 시행
- MS 데이터에서에서 다중 집단 분석 시행
- MS 데이터 분석의 시각화
-
- 8장 차세대 시퀀싱 데이터 분석
- 소개
- SRA 데이터베이스 쿼리
- SRA 데이터베이스로부터 데이터 다운로드
- R에서 FASTQ 파일 읽기
- 정렬 데이터 읽기
- 차세대 시퀀싱 원 데이터 전처리
- edgeR 패키지를 사용한 RNAseq 데이터 분석
- limma를 사용한 시퀀싱 데이터의 층별 분석
- 유전자 온톨로지 용어를 사용한 RNAseq 데이터 농축
- 서열 데이터의 KEGG 농축
- 메틸화 데이터의 분석
- ChipSeq 데이터 분석
- 차세대 시퀀싱 데이터 시각화
- 9장 생명정보학에서의 기계 학습
- 소개
- k-means와 계층적 군집을 이용한 데이터 군집화
- 군집의 시각화
- 분류에 대한 지도 학습
- Naïve Bayes를 이용한 확률적 학습
- 기계 학습에서 부트스트래핑
- 분류기에 대한 교차 타당화
- 분류기의 성능 평가
- ROC 커브의 시각화
- 어레이 데이터를 이용한 생체지표 확인
- 부록A. R에서 유용한 연산자와 함수
- 부록B. 유용한 R 패키지
도서 오류 신고
정오표
정오표
p4
독일의 프라운호퍼 알고리즘과 과학 컴퓨팅 연구소
->
독일에 위치한 알고리즘과 과학 컴퓨팅 프라운호퍼 연구소
p21
https://www.packtpub.com/sites/default/files/downloads/31320S_ColoredImages.pdf
->
https://www.packtpub.com/sites/default/files/downloads/3132OS_ColoredImages.pdf
p49 그림 수정